Il MIT utilizza l’intelligenza artificiale per identificare nuovi potenti antibiotici
Di ANNE TRAFTON, MASSACHUSETTS INSTITUTE OF TECHNOLOGY
Un modello di apprendimento profondo identifica un nuovo potente farmaco in grado di uccidere molte specie di batteri resistenti agli antibiotici.
Utilizzando un algoritmo di apprendimento automatico, i ricercatori del MIT hanno identificato un nuovo potente composto antibiotico. In test di laboratorio, il farmaco ha ucciso molti dei batteri più problematici che causano malattie, tra cui alcuni ceppi resistenti a tutti gli antibiotici noti. Ha anche eliminato le infezioni in due diversi modelli di topo.
Il modello computerizzato, che può selezionare più di cento milioni di composti chimici in pochi giorni, è progettato per individuare potenziali antibiotici che uccidono i batteri utilizzando meccanismi diversi rispetto a quelli dei farmaci esistenti.
“Volevamo sviluppare una piattaforma che ci consentisse di sfruttare il potere dell’intelligenza artificiale per inaugurare una nuova era di scoperta di farmaci antibiotici”, afferma James Collins, professore di ingegneria medica e scienze presso l’Istituto di ingegneria e scienza del MIT (IMES) e Dipartimento di Ingegneria Biologica. “Il nostro approccio ha rivelato questa straordinaria molecola che è probabilmente uno degli antibiotici più potenti che è stato scoperto.”
Nel loro nuovo studio, i ricercatori hanno anche identificato molti altri promettenti candidati antibiotici, che hanno intenzione di testare ulteriormente. Ritengono che il modello potrebbe anche essere utilizzato per progettare nuovi farmaci, sulla base di ciò che ha appreso sulle strutture chimiche che consentono ai farmaci di uccidere i batteri.
“Il modello di apprendimento automatico può esplorare, in silico, ampi spazi chimici che possono essere proibitivi per gli approcci sperimentali tradizionali”, afferma Regina Barzilay, professore Delta Electronics di ingegneria elettrica e informatica nel laboratorio di informatica e intelligenza artificiale del MIT (CSAIL) .

Barzilay e Collins, che sono co-lead della facoltà per la clinica Abdul Latif Jameel del MIT per l’apprendimento automatico in salute, sono gli autori senior dello studio, che appare oggi su Cell . Il primo autore dell’articolo è Jonathan Stokes, postdoc presso il MIT e il Broad Institute of MIT e Harvard.
Una nuova pipeline
Negli ultimi decenni, sono stati sviluppati pochissimi nuovi antibiotici e la maggior parte di quelli recentemente approvati sono varianti leggermente diverse dei farmaci esistenti. Gli attuali metodi per lo screening di nuovi antibiotici sono spesso proibitivi in termini di costi, richiedono un investimento significativo in termini di tempo e sono generalmente limitati a uno spettro ristretto di diversità chimica.
“Stiamo affrontando una crescente crisi intorno alla resistenza agli antibiotici, e questa situazione è generata sia da un numero crescente di agenti patogeni che diventano resistenti agli antibiotici esistenti, sia da una conduttura anemica nelle industrie biotecnologiche e farmaceutiche per i nuovi antibiotici”, afferma Collins.
Per cercare di trovare composti completamente nuovi, ha collaborato con Barzilay, il professor Tommi Jaakkola e i loro studenti Kevin Yang, Kyle Swanson e Wengong Jin, che hanno precedentemente sviluppato modelli di computer di apprendimento automatico che possono essere addestrati per analizzare le strutture molecolari di i composti e li correlano con tratti particolari, come la capacità di uccidere i batteri.
L’idea di utilizzare modelli computerizzati predittivi per lo screening “in silico” non è nuova, ma fino ad ora questi modelli non erano sufficientemente accurati per trasformare la scoperta di droghe. In precedenza, le molecole erano rappresentate come vettori che riflettevano la presenza o l’assenza di determinati gruppi chimici. Tuttavia, le nuove reti neurali possono apprendere automaticamente queste rappresentazioni, mappando le molecole in vettori continui che vengono successivamente utilizzati per prevederne le proprietà.
In questo caso, i ricercatori hanno progettato il loro modello per cercare caratteristiche chimiche che rendano le molecole efficaci nell’uccidere E. coli. Per fare ciò, hanno addestrato il modello su circa 2.500 molecole, tra cui circa 1.700 farmaci approvati dalla FDA e un set di 800 prodotti naturali con diverse strutture e una vasta gamma di bioattività.
Una volta che il modello è stato addestrato, i ricercatori lo hanno testato sull’Hub Repurposing Hub del Broad Institute, una biblioteca di circa 6.000 composti. Il modello ha individuato una molecola che aveva una forte attività antibatterica e una struttura chimica diversa da qualsiasi antibiotico esistente. Utilizzando un diverso modello di apprendimento automatico, i ricercatori hanno anche dimostrato che questa molecola avrebbe probabilmente una bassa tossicità per le cellule umane.
Questa molecola, che i ricercatori hanno deciso di chiamare halicin, dopo il sistema di intelligenza artificiale immaginaria di “2001: Odissea nello spazio”, è stata precedentemente studiata come possibile farmaco per il diabete. I ricercatori lo hanno testato contro dozzine di ceppi batterici isolati da pazienti e cresciuti in piatti di laboratorio e hanno scoperto che era in grado di uccidere molti che sono resistenti al trattamento, tra cui Clostridium difficile, Acinetobacter baumannii e Mycobacterium tuberculosis. Il farmaco ha funzionato contro ogni specie testata, ad eccezione di Pseudomonas aeruginosa, un patogeno polmonare difficile da trattare.
Per testare l’efficacia dell’alicina negli animali vivi, i ricercatori l’hanno usato per trattare i topi infetti da A. baumannii, un batterio che ha infettato molti soldati statunitensi di stanza in Iraq e Afghanistan. Il ceppo di A. baumannii che hanno usato è resistente a tutti gli antibiotici noti, ma l’applicazione di un unguento contenente allicina ha eliminato completamente le infezioni entro 24 ore.
Studi preliminari suggeriscono che l’alicina uccide i batteri interrompendo la loro capacità di mantenere un gradiente elettrochimico attraverso le loro membrane cellulari. Questo gradiente è necessario, tra le altre funzioni, per produrre ATP (molecole che le cellule usano per immagazzinare energia), quindi se il gradiente si rompe, le cellule muoiono. Questo tipo di meccanismo di uccisione potrebbe essere difficile per i batteri sviluppare resistenza, affermano i ricercatori.
“Quando hai a che fare con una molecola che probabilmente si associa ai componenti della membrana, una cellula non può necessariamente acquisire una singola mutazione o un paio di mutazioni per cambiare la chimica della membrana esterna. Mutazioni come questa tendono ad essere molto più complesse da acquisire in modo evolutivo ”, afferma Stokes.
In questo studio, i ricercatori hanno scoperto che E. coli non ha sviluppato alcuna resistenza all’alicina durante un periodo di trattamento di 30 giorni. Al contrario, i batteri hanno iniziato a sviluppare resistenza all’antibiotico ciprofloxacina entro uno o tre giorni e dopo 30 giorni i batteri erano circa 200 volte più resistenti alla ciprofloxacina di quanto non fossero all’inizio dell’esperimento.
I ricercatori hanno in programma di proseguire ulteriori studi sull’alicina, lavorando con un’azienda farmaceutica o un’organizzazione senza scopo di lucro, nella speranza di svilupparlo per l’uso nell’uomo.
Molecole ottimizzate
Dopo aver identificato l’alicina, i ricercatori hanno anche usato il loro modello per selezionare oltre 100 milioni di molecole selezionate dal database ZINC15, una raccolta online di circa 1,5 miliardi di composti chimici. Questo schermo, che ha richiesto solo tre giorni, ha identificato 23 candidati strutturalmente diversi dagli antibiotici esistenti e previsti non tossici per le cellule umane.
Nei test di laboratorio contro cinque specie di batteri, i ricercatori hanno scoperto che otto delle molecole hanno mostrato attività antibatterica e due erano particolarmente potenti. I ricercatori hanno ora in programma di testare ulteriormente queste molecole e anche di esaminare più del database ZINC15.
I ricercatori hanno anche in programma di utilizzare il loro modello per progettare nuovi antibiotici e ottimizzare le molecole esistenti. Ad esempio, potrebbero addestrare il modello ad aggiungere funzionalità che potrebbero indirizzare un determinato antibiotico solo su determinati batteri, impedendogli di uccidere i batteri benefici nel tratto digestivo di un paziente.
Riferimento: “Un approccio di apprendimento profondo alla scoperta di antibiotici” di Jonathan M. Stokes, Kevin Yang, Kyle Swanson, Wengong Jin, Andres Cubillos-Ruiz, Nina M. Donghia, Craig R. MacNair, Shawn French, Lindsey A. Carfrae, Zohar Bloom-Ackerman, Victoria M. Tran, Anush Chiappino-Pepe, Ahmed H. Badran, Ian W. Andrews, Emma J. Chory, George M. Church, Eric D. Brown, Tommi S. Jaakkola, Regina Barzilay e James J. Collins
La ricerca è stata finanziata dalla Abdul Latif Jameel Clinic for Machine Learning in Health, dalla Defense Threat Reduction Agency, dal Broad Institute, dal DARPA Make-It Program, dal Canadian Institutes of Health Research, dalla Canadian Foundation for Innovation, dal Canada Research Chairs Programma, Banting Fellowships Program, Human Frontier Science Program, Pershing Square Foundation, Swiss National Science Foundation, premio National Institutes of Health Early Investigator, National Science Foundation Graduate Research Fellowship Program e un regalo di Anita e Josh Bekenstein .
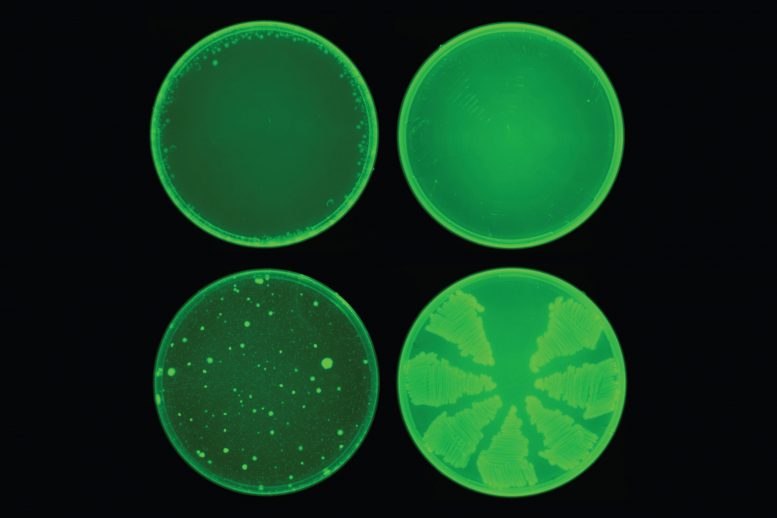
AI Nuovo antibiotico
I ricercatori del MIT hanno utilizzato un algoritmo di apprendimento automatico per identificare un farmaco chiamato halicin che uccide molti ceppi di batteri. L’alicina (fila in alto) ha impedito lo sviluppo di resistenza agli antibiotici in E. coli, mentre la ciprofloxacina (fila in basso) no. Credito: per gentile concessione del Collins Lab presso il MIT
Antibiotico di intelligenza artificiale
I ricercatori del MIT hanno identificato un nuovo potente composto antibiotico usando un algoritmo di apprendimento automatico.