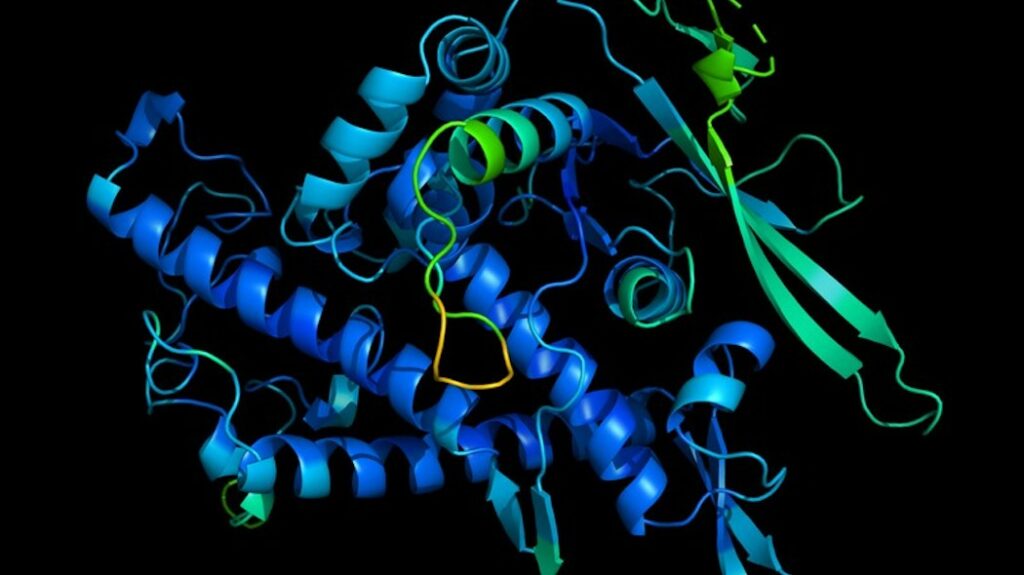
La previsione migliorata del ripiegamento delle proteine di DeepMind potrebbe accelerare la scoperta di farmaci
Quando si tratta delle aspettative dei clienti, la pandemia ha cambiato tutto
Scopri come accelerare il servizio clienti, ottimizzare i costi e migliorare il self-service in un mondo incentrato sul digitale.
La ricetta per le proteine - grandi molecole costituite da aminoacidi che sono i mattoni fondamentali di tessuti, muscoli, capelli, enzimi, anticorpi e altre parti essenziali degli organismi viventi – sono codificate nel DNA. Sono queste definizioni genetiche che circoscrivono le loro strutture tridimensionali, che a loro volta determina le loro capacità. Ma il “ripiegamento” delle proteine, come viene chiamato, è notoriamente difficile da capire solo da una sequenza genetica corrispondente. Il DNA contiene solo informazioni sulle catene di residui di amminoacidi e non sulla forma finale di tali catene.
Nel dicembre 2018, DeepMind ha tentato di affrontare la sfida del ripiegamento delle proteine con un sistema di apprendimento automatico chiamato AlphaFold. Frutto di due anni di lavoro, la sussidiaria di Alphabet disse all’epoca che AlphaFold poteva prevedere le strutture in modo più preciso rispetto alle soluzioni precedenti. Dando credito a questa affermazione, il sistema ha battuto 98 concorrenti nella competizione di ripiegamento delle proteine CASP (Critical Assessment of Structure Prediction) a Cancun, dove ha previsto con successo la struttura di 25 proteine su 43.
DeepMind ora afferma che AlphaFold ha superato per la seconda volta i metodi di previsione del ripiegamento delle proteine concorrenti. Nei risultati della 14a valutazione CASP, una versione più recente di AlphaFold – AlphaFold 2 – ha un errore medio paragonabile alla larghezza di un atomo (o 0,1 di un nanometro), competitiva con i risultati dei metodi sperimentali.
“Siamo rimasti bloccati su questo problema – come si ripiegano le proteine - per quasi 50 anni”, ha detto ai giornalisti la scorsa settimana il professore dell’Università del Maryland John Moult, cofondatore e presidente del CASP. “Vedere DeepMind produrre una soluzione per questo, avendo lavorato personalmente su questo problema per così tanto tempo e dopo così tante fermate e partenze, chiedendoci se saremmo mai arrivati lì, è un momento molto speciale.”
Ripiegamento delle proteine
Le soluzioni a molte delle sfide del mondo, come lo sviluppo di trattamenti per le malattie, possono essere ricondotte alle proteine. Le proteine anticorpali hanno la forma di una “Y”, ad esempio, consentendo loro di attaccarsi a virus e batteri, e le proteine del collagene hanno la forma di corde, che trasmettono la tensione tra cartilagine, ossa, pelle e legamenti. In SARS-CoV-2, il nuovo coronavirus, una proteina simile a un picco cambia forma per interagire con un’altra proteina sulla superficie delle cellule umane, consentendole di forzare l’ingresso.
Fu il biochimico Christian Anfinsen a ipotizzare nel 1972 che la sequenza di amminoacidi di una proteina potesse determinarne la struttura. Ciò ha posto le basi per i tentativi di prevedere la struttura di una proteina in base alla sua sequenza di amminoacidi come alternativa a metodi sperimentali costosi e che richiedono tempo come la risonanza magnetica nucleare, la cristallografia a raggi X e la microscopia crioelettronica. A complicare le cose, tuttavia, è la pura complessità del ripiegamento delle proteine. Gli scienziati stimano che a causa del numero incalcolabile di interazioni tra gli amminoacidi, ci vorrebbero più di 13,8 miliardi di anni per capire tutte le possibili configurazioni di una tipica proteina prima di identificare la giusta struttura.
DeepMind afferma che il suo approccio con AlphaFold trae ispirazione dai campi della biologia, della fisica, dell’apprendimento delle macchine e del lavoro degli scienziati nell’ultimo mezzo secolo. Approfittando del fatto che una proteina ripiegata può essere pensata come un “grafico spaziale”, dove i residui di amminoacidi (aminoacidi contenuti in un peptide o una proteina) sono nodi e i bordi collegano i residui in stretta vicinanza, AlphaFold sfrutta un algoritmo AI che tenta di interpretare la struttura di questo grafico mentre ragiona sul grafico implicito che sta costruendo usando sequenze evolutivamente correlate, allineamento di sequenze multiple e una rappresentazione di coppie di residui di amminoacidi.
Ripetendo questo processo, AlphaFold può imparare a prevedere la struttura sottostante di una proteina e determinarne la forma in pochi giorni, secondo DeepMind. Inoltre, il sistema può auto-valutare quali parti di ciascuna struttura proteica sono affidabili utilizzando una misura di confidenza interna.
DeepMind afferma che la versione più recente di AlphaFold, che sarà descritta in dettaglio in un articolo di prossima pubblicazione, è stata addestrata su circa 170.000 strutture proteiche dalla Protein Data Bank, un database open source per i dati strutturali di grandi molecole biologiche. L’azienda ha sfruttato 128 unità di elaborazione tensoriale (TPU) di terza generazione di Google, chip acceleratori AI speciali disponibili tramite Google Cloud, per risorse di calcolo equivalenti a circa 100-200 schede grafiche. La formazione ha richiesto alcune settimane. Per motivi di confronto, ci sono voluti DeepMind 44 giorni per formare un singolo agente nel suo 2-gioco StarCraft AlphaStar sistema usando 32 terza generazione TPU.
DeepMind ha rifiutato di rivelare il costo dell’addestramento di AlphaFold. Ma Google addebita ai clienti di Google Cloud $ 32 l’ora per TPU di terza generazione, che equivale a circa $ 688.128 a settimana.
Nel 1994, Moult e l’Università della California, il professore di Davis Krzysztof Fidelis hanno fondato CASP come valutazione cieca biennale per catalizzare la ricerca, monitorare i progressi e stabilire lo stato dell’arte nella previsione della struttura delle proteine. È considerato il gold standard per il benchmarking delle tecniche predittive, perché CASP sceglie strutture che sono state selezionate solo di recente sperimentalmente come obiettivi per i team per testare i loro metodi di previsione. Alcuni erano ancora in attesa di convalida al momento della valutazione di AlphaFold.
Poiché le strutture target non vengono pubblicate in anticipo, i partecipanti al CASP devono prevedere ciecamente la struttura di ciascuna delle proteine. Queste previsioni vengono quindi confrontate con i dati sperimentali della verità fondamentale quando questi dati diventano disponibili.
La metrica principale utilizzata da CASP per misurare l’accuratezza delle previsioni è il test della distanza globale, che varia da 0 a 100. È essenzialmente la percentuale di residui di amminoacidi entro una certa distanza di soglia dalla posizione corretta. Un punteggio di circa 90 è considerato informalmente competitivo con i risultati ottenuti da metodi sperimentali; AlphaFold ha ottenuto un punteggio mediano di 92,4 complessivi e un punteggio mediano di 87 per le proteine nella categoria di modellazione libera (cioè, quelle senza modelli).
“Quello che abbiamo visto in CASP14 è stato un gruppo che ha fornito precisione atomica”, ha detto Moult. “Questo [progresso] ti dà una tale eccitazione per il modo in cui funziona la scienza – su come non puoi mai vedere esattamente, o anche approssimativamente, cosa succederà dopo. Ci sono sempre queste sorprese. E questo in realtà come scienziato è ciò che ti fa andare avanti. Quale sarà la prossima sorpresa? “
Applicazioni del mondo reale
DeepMind sostiene che AlphaFold, se ulteriormente perfezionato, potrebbe essere applicato a problemi precedentemente intrattabili nel campo del ripiegamento delle proteine, compresi quelli relativi agli sforzi epidemiologici. All’inizio di quest’anno, la società aveva previsto diverse strutture proteiche di SARS-CoV-2, tra cui ORF3a, la cui composizione era in precedenza un mistero. Al CASP14, DeepMind ha previsto la struttura di un’altra proteina del coronavirus, ORF8, che da allora è stata confermata dagli sperimentatori.
Al di là della risposta alla pandemia, DeepMind prevede che AlphaFold sarà utilizzato per esplorare le centinaia di milioni di proteine per le quali la scienza attualmente manca di modelli. Poiché il DNA specifica le sequenze di amminoacidi che comprendono le strutture proteiche, i progressi della genomica hanno reso possibile la lettura di sequenze proteiche dal mondo naturale, con 180 milioni di sequenze proteiche e conteggio nel database Universal Protein disponibile al pubblico. Al contrario, dato il lavoro sperimentale necessario per tradurre da sequenza a struttura, solo circa 170.000 strutture proteiche si trovano nella Protein Data Bank.
DeepMind afferma di essere impegnata a rendere AlphaFold disponibile “su larga scala” e a collaborare con i partner per esplorare nuove frontiere, come il modo in cui più proteine formano complessi e interagiscono con DNA, RNA e piccole molecole. Migliorare la comprensione della comunità scientifica del ripiegamento delle proteine potrebbe portare a diagnosi e cure più efficaci di malattie come il Parkinson e l’Alzheimer, poiché si ritiene che queste siano causate da proteine mal ripiegate. E potrebbe aiutare nella progettazione delle proteine, portando a batteri che secernono proteine che rendono le acque reflue biodegradabili, ad esempio, ed enzimi che possono aiutare a gestire inquinanti come la plastica e il petrolio.
In ogni caso, è una pietra miliare per DeepMind, il cui lavoro si è concentrato principalmente sul dominio dei giochi. Il suo sistema AlphaStar ha battuto i giocatori professionisti di StarCraft 2, dopo le vittorie di AlphaZero su Go, scacchi e shogi. Mentre alcuni dei lavori di DeepMind hanno trovato applicazioni nel mondo reale, principalmente nei data center , nelle auto a guida autonoma di Waymo e negli algoritmi di raccomandazione del Google Play Store , DeepMind deve ancora raggiungere un significativo passo avanti con l’IA in un’area scientifica come il ripiegamento delle proteine o la dinamica del vetro modellazione . Questi nuovi risultati potrebbero segnare un cambiamento nelle fortune dell’azienda.
“AlphaFold rappresenta un enorme balzo in avanti che spero possa davvero accelerare la scoperta di farmaci e aiutarci a comprendere meglio la malattia. È piuttosto strabiliante “, ha detto il CEO di DeepMind Demis Hassabis durante il briefing della scorsa settimana. “Abbiamo avanzato lo stato dell’arte nel campo, quindi è fantastico, ma c’è ancora molta strada da fare prima di risolverlo.”