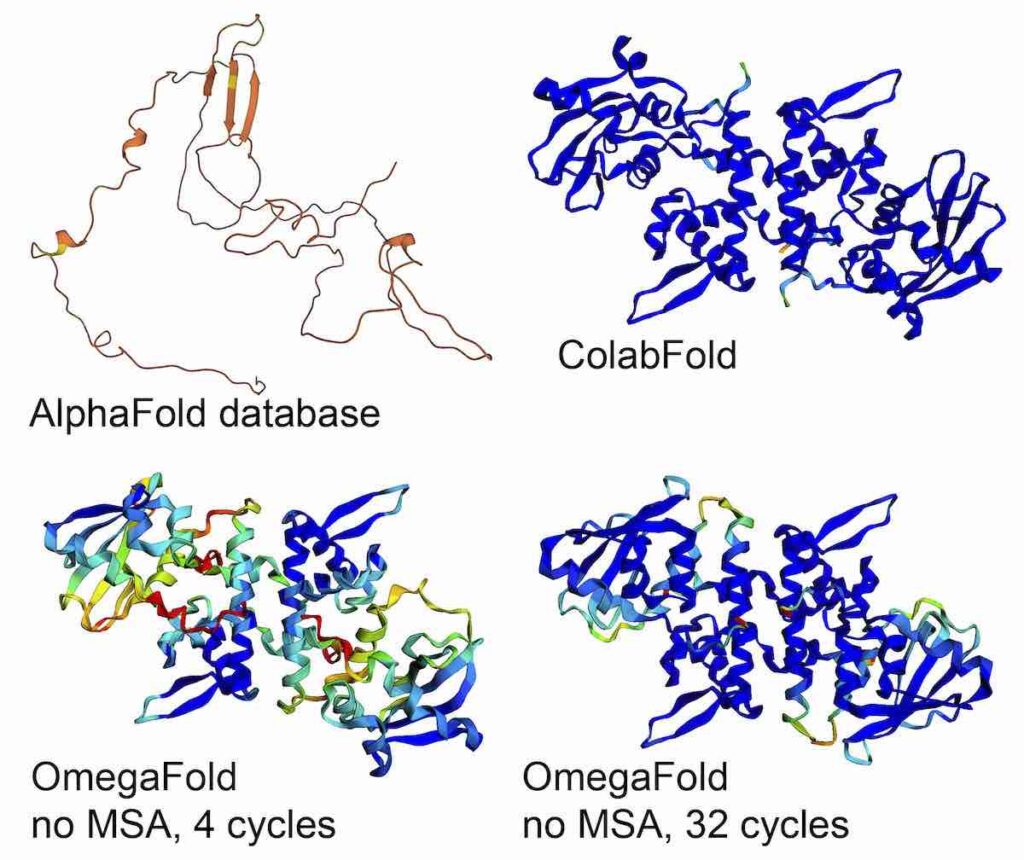
OmegaFold ha ottenuto una precisione di previsione statistica molto più elevata rispetto ad AlphaFold 2
Il 20 luglio 2022, l’azienda biotecnologica cinese Helixon ha lanciato OmegaFold, il primo metodo computazionale per prevedere con successo la struttura proteica ad alta risoluzione da una singola sequenza primaria. Questo nuovo studio condotto da ricercatori cinesi colma una lacuna molto riscontrata nella previsione della struttura e si avvicina a pochi centimetri dalla comprensione del ripiegamento delle proteine in natura.
Di recente, la società ha reso open source il suo progetto, unendosi ad altri come AlphaFold di DeepMind , RoseTTAFold e ESMFold di Meta AI , anch’essi open source. La versione iniziale del codice e del modello è disponibile su GitHub .
Comprendere il ripiegamento delle proteine aiuta ricercatori e scienziati a conoscere la causa alla base di molte malattie e anomalie. Aiuta anche a trovare una cura, progettare nuovi farmaci, soluzioni farmaceutiche e trattamenti alternativi.
Questo nuovo modello sviluppato da Helixon afferma di superare RoseTTAFold e ottenere una precisione di previsione simile a AlphaFold 2 sulla struttura rilasciata di recente. In uno studio , i ricercatori hanno affermato di aver utilizzato una nuova combinazione di un modello di linguaggio proteico che consente loro di fare previsioni da singole sequenze e un modello trasformatore ispirato alla geometria addestrato su strutture proteiche.
Inoltre, OmegaFold consente previsioni accurate su proteine orfane che non appartengono ad alcuna famiglia di proteine caratterizzate dalla funzionalità e anticorpi che tendono ad avere MSA (allineamenti di sequenze multiple) rumorosi a causa della rapida evoluzione.
OmegaFold vs AlphaFold vs ESMfold
Un mese fa, Meta AI ha lanciato un modello rivoluzionario chiamato Evolutionary Scale Modeling , o ESM, per una previsione più rapida della struttura delle proteine. Anche questo modello ha affermato di avere un’accuratezza simile a quella di AlphaFold2 e RoseTTAFold, ma l’inferenza di ESMFold è più veloce nel consentire l’esplorazione degli spazi strutturali delle proteine metagenomiche .
Sembrano esserci evidenti somiglianze tra ESMfold, AlphaFold e OmegaFold. Il team ha affermato che il modello generale di OmegaFold è concettualmente ispirato dai progressi nei modelli linguistici per la NLP accoppiati con le reti neurali profonde utilizzate in AlphaFold2.
OmegaFold sfrutta un modello di linguaggio proteico basato su trasformatore profondo , addestrato su un’ampia raccolta di sequenze proteiche non allineate e non etichettate, per apprendere le rappresentazioni di residui singoli e a coppie come potenti funzionalità che modellano la distribuzione delle sequenze.
Il modello del linguaggio della proteina Omega (PLM) può acquisire informazioni strutturali e funzionali codificate nelle sequenze di amminoacidi attraverso gli incorporamenti. Questi vengono successivamente inseriti in Geoformer , una nuova rete neurale del trasformatore ispirata alla geometria , per distillare le relazioni strutturali e fisiche a coppie tra gli amminoacidi. Infine, un modulo strutturale prevede le coordinate 3D di tutti gli atomi pesanti.
ESMfold , d’altra parte, sfrutta un modello linguistico su larga scala per la previsione delle proteine. I miglioramenti nella perplessità della modellazione linguistica e nell’apprendimento della struttura continuano attraverso 15 miliardi di parametri. Nel frattempo, AlphaFold utilizza un’architettura basata sulla rete e i proventi della formazione basati su vincoli evolutivi, fisici e geometrici delle strutture proteiche.
I ricercatori hanno notato che il loro modello (OmegaFold) funziona bene sui set di dati di riferimento CASP e CAMEO, coprendo un’ampia gamma di livelli di difficoltà di previsione. In confronto, OmegaFold, con una singola sequenza come input, era accurato quanto i metodi avanzati basati su MSA, inclusi AlphaFold 2 e RoseTTATold.
Come mostrato di seguito, le strutture OmegaFold avevano un punteggio medio del test di differenza di distanza locale (LDDT) di 0,82 sul set di dati CAMEO, con accuratezza comparabile alle strutture RoseTTAFold (punteggio LDDT medio 0,75) e simile alle strutture AlphaFold 2 (LDDT medio 0,86) previsto da MSA. I test di differenza a distanza locale, o LDDT, sono metriche comunemente utilizzate per la valutazione della struttura.
Sul set di dati CASP, anche le strutture OmegaFold erano abbastanza accurate, con un punteggio TM medio di 0,79, leggermente inferiore a quello delle strutture RoseTTAFold (punteggio TM medio 0,81) ed equivalente alle strutture AlphaFold 2 (punteggio TM medio 0,79). Nel frattempo, ESMFold ha ottenuto un punteggio TM di 0,71 sul set di test CAMEO e 0,53 sul set di dati CASP. Il punteggio TM è una metrica comune per valutare la somiglianza topologica della struttura delle proteine.
Un punteggio superiore a 0,90 è considerato più o meno equivalente alla struttura determinata sperimentalmente.
Su input a sequenza singola, OmegaFold vince
Nel corso degli anni, diverse aziende hanno utilizzato il deep learning per sfruttare le informazioni evolutive negli MSA (allineamenti di sequenze multiple) per prevedere con precisione le strutture delle proteine. Al contrario, gli MSA delle proteine omologhe non sono sempre disponibili, comprese le proteine orfane e gli anticorpi, e una proteina si ripiega tipicamente in un ambiente naturale dalla sua sequenza amminoacidica primaria nella sua struttura 3D. Il team di OmegaFold ha suggerito che le informazioni evolutive e gli MSA non dovrebbero essere necessari per prevedere la forma ripiegata di una proteina.
È qui che entra in gioco il nuovo modello di produzione proteica “super veloce” OmegaFold. Ha superato AlphaFold 2 e RoseTTAFold sugli ingressi a sequenza singola. Inoltre, OmegaFold ha ottenuto un’accuratezza di previsione statistica molto più elevata rispetto ad AlphaFold 2, probabilmente grazie ai vantaggi del suo metodo di previsione basato su sequenza singola, sia sui loop di anticorpi che sulle proteine orfane.