Andare oltre il cervello umano: il deep learning assume la biologia sintetica
Il lavoro del membro della Wyss Core Faculty Peng Yin in collaborazione con Collins e altri ha dimostrato che è possibile combinare diversi interruttori di appoggio per calcolare la presenza di più “trigger”, in modo simile alla scheda logica di un computer.
Il DNA e l’ RNA sono stati paragonati a “manuali di istruzioni” contenenti le informazioni necessarie al funzionamento delle “macchine” viventi. Ma mentre le macchine elettroniche come computer e robot sono progettate da zero per servire uno scopo specifico, gli organismi biologici sono governati da un insieme di funzioni molto più disordinato e complesso che non ha la prevedibilità del codice binario. Inventare nuove soluzioni ai problemi biologici richiede di prendere in considerazione variabili apparentemente intrattabili, un compito che è scoraggiante anche per i cervelli umani più intrepidi.
Due team di scienziati del Wyss Institute dell’Università di Harvard e del Massachusetts Institute of Technology hanno ideato percorsi per aggirare questo ostacolo andando oltre il cervello umano; hanno sviluppato una serie di algoritmi di apprendimento automatico in grado di analizzare risme di sequenze “toehold” basate su RNA e prevedere quali saranno più efficaci nel rilevare e rispondere a una sequenza target desiderata. Come riportato in due articoli pubblicati contemporaneamente oggi (7 ottobre 2020) su Nature Communications , gli algoritmi potrebbero essere generalizzabili anche ad altri problemi della biologia sintetica e potrebbero accelerare lo sviluppo di strumenti biotecnologici per migliorare la scienza e la medicina e aiutare a salvare vite umane.
“Questi risultati sono entusiasmanti perché segnano il punto di partenza della nostra capacità di porre domande migliori sui principi fondamentali del ripiegamento dell’RNA, che dobbiamo conoscere al fine di ottenere scoperte significative e costruire tecnologie biologiche utili”, ha affermato Luis Soenksen, Ph. D., postdoctoral Fellow presso il Wyss Institute e Venture Builder presso la Jameel Clinic del MIT , co-primo autore del primo dei due articoli.
In questa animazione, il postdoctoral Fellow del Wyss Institute Alex Green, Ph.D., l’autore principale di “Toehold Switches: De-Novo-Designed Regulators of Gene Expression”, racconta una guida passo passo al meccanismo del toehold sintetico switch gene regolatore. Crediti: Wyss Institute presso Harvard University
Controllo degli interruttori in punta
La collaborazione tra i data scientist della Predictive BioAnalytics Initiative del Wyss Institute e i biologi sintetici nel laboratorio del MIT Jim Collins, membro della Wyss Core Faculty, è stata creata per applicare la potenza di calcolo dell’apprendimento automatico, delle reti neurali e di altre architetture algoritmiche a problemi complessi in biologia che finora hanno sfidato la risoluzione. Come terreno di prova per il loro approccio, i due team si sono concentrati su una classe specifica di molecole di RNA ingegnerizzate: gli interruttori di blocco, che sono piegati in una forma a forcina nel loro stato “spento”. Quando un filamento di RNA complementare si lega a una sequenza “trigger” che scende da un’estremità della forcina, l’interruttore del puntale si apre nel suo stato “on” ed espone sequenze che erano precedentemente nascoste all’interno della forcina consentendo ai ribosomi di legarsi e tradurre un gene a valle in molecole proteiche. Questo controllo preciso sull’espressione dei geni in risposta alla presenza di una determinata molecola rende gli interruttori a contatto con componenti molto potenti per rilevare le sostanze nell’ambiente, rilevare malattie e altri scopi.
Tuttavia, molti interruttori toehold non funzionano molto bene se testati sperimentalmente, anche se sono stati progettati per produrre un output desiderato in risposta a un dato input basato su regole di ripiegamento dell’RNA note. Riconoscendo questo problema, i team hanno deciso di utilizzare l’apprendimento automatico per analizzare un grande volume di sequenze di interruttori dei piedi e utilizzare le intuizioni di tale analisi per prevedere in modo più accurato quali appigli eseguono in modo affidabile i compiti previsti, il che consentirebbe ai ricercatori di identificare rapidamente appigli di alta qualità per vari esperimenti.
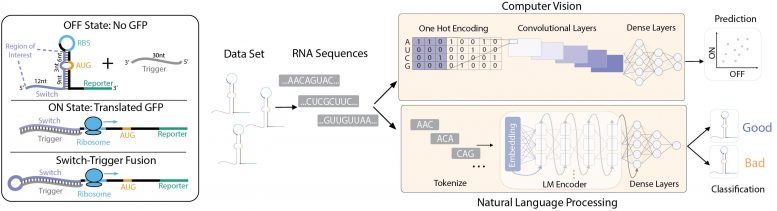
Deep Learning Framework RNA
Dopo aver generato un set di dati di migliaia di interruttori toehold, un team ha utilizzato un algoritmo basato sulla visione artificiale per analizzare le sequenze toehold come immagini bidimensionali, mentre l’altro team ha utilizzato l’elaborazione del linguaggio naturale per interpretare le sequenze come “parole” scritte nel “Linguaggio” di RNA. Crediti: Wyss Institute presso Harvard University
Il primo ostacolo che hanno dovuto affrontare è stato il fatto che non esisteva un set di dati di sequenze di interruttori a leva sufficientemente grande da consentire un’analisi efficace delle tecniche di apprendimento profondo. Gli autori si sono impegnati a generare un set di dati che sarebbe stato utile per addestrare tali modelli. “Abbiamo progettato e sintetizzato un’enorme libreria di interruttori a contatto, quasi 100.000 in totale, campionando sistematicamente regioni di trigger brevi lungo l’intero genoma di 23 virus e 906 fattori di trascrizione umani”, ha affermato Alex Garruss, uno studente laureato di Harvard che lavora presso il Wyss Institute chi è un co-primo autore del primo articolo. “La scala senza precedenti di questo set di dati consente l’uso di tecniche avanzate di apprendimento automatico per identificare e comprendere interruttori utili per applicazioni a valle immediate e progettazione futura”.
Armati di dati sufficienti, i team hanno utilizzato per la prima volta gli strumenti tradizionalmente utilizzati per analizzare le molecole di RNA sintetico per vedere se potevano prevedere con precisione il comportamento degli interruttori a leva ora che erano disponibili molti più esempi. Tuttavia, nessuno dei metodi che hanno provato, compresa la modellazione meccanicistica basata sulla termodinamica e sulle caratteristiche fisiche, è stato in grado di prevedere con sufficiente precisione quali appigli funzionassero meglio.
Un’immagine vale mille paia di basi
I ricercatori hanno quindi esplorato varie tecniche di apprendimento automatico per vedere se potevano creare modelli con migliori capacità predittive. Gli autori del primo articolo hanno deciso di analizzare gli interruttori di appoggio non come sequenze di basi, ma piuttosto come “immagini” bidimensionali di possibilità di coppie di basi. “Conosciamo le regole di base per il modo in cui le coppie di basi di una molecola di RNA si legano tra loro, ma le molecole sono sinuose: non hanno mai una singola forma perfetta, ma piuttosto una probabilità di forme diverse in cui potrebbero trovarsi”, ha detto Nicolaas Angenent-Mari, uno studente laureato del MIT che lavora al Wyss Institute e co-primo autore del primo articolo. “Gli algoritmi di visione artificiale sono diventati molto bravi nell’analisi delle immagini, quindi abbiamo creato una rappresentazione simile a un’immagine di tutti i possibili stati di piegatura di ciascun interruttore del piede,

Modelli di framework di deep learning
Utilizzando entrambi i modelli in sequenza, i ricercatori sono stati in grado di prevedere quali sequenze di appoggio avrebbero prodotto sensori di alta qualità. Crediti: Wyss Institute presso Harvard University
Un altro vantaggio del loro approccio basato sulla visualizzazione è che il team è stato in grado di “vedere” a quali parti di una sequenza di interruttori in appiglio l’algoritmo ha “prestato maggiore attenzione” nel determinare se una determinata sequenza fosse “buona” o “cattiva”. Hanno chiamato questo approccio interpretativo Visualizing Secondary Structure Saliency Maps, o VIS4Map, e lo hanno applicato all’intero set di dati dell’interruttore toehold. VIS4Map ha identificato con successo gli elementi fisici degli interruttori del toehold che hanno influenzato le loro prestazioni e ha permesso ai ricercatori di concludere che i toehold con strutture interne più potenzialmente concorrenti erano “più deboli” e quindi di qualità inferiore rispetto a quelli con un minor numero di tali strutture, fornendo informazioni sui meccanismi di piegatura dell’RNA che non era stato scoperto utilizzando tecniche di analisi tradizionali.
“Essere in grado di capire e spiegare perché determinati strumenti funzionano o non funzionano è stato un obiettivo secondario all’interno della comunità dell’intelligenza artificiale per un po ‘di tempo, ma l’interpretabilità deve essere al centro delle nostre preoccupazioni quando si studia la biologia perché le ragioni alla base di questi i comportamenti dei sistemi spesso non possono essere intuiti “, ha detto Jim Collins, Ph.D., l’autore senior del primo articolo. “Scoperte e interruzioni significative sono il risultato di una profonda comprensione di come funziona la natura e questo progetto dimostra che l’apprendimento automatico, se progettato e applicato correttamente, può migliorare notevolmente la nostra capacità di ottenere informazioni importanti sui sistemi biologici”. Collins è anche il Termeer Professor di ingegneria medica e scienza al MIT.
Adesso parli la mia lingua
Mentre il primo team ha analizzato le sequenze di interruttori del piede come immagini 2D per prevederne la qualità, il secondo team ha creato due diverse architetture di apprendimento profondo che hanno affrontato la sfida utilizzando tecniche ortogonali. Sono poi andati oltre la previsione della qualità del toehold e hanno utilizzato i loro modelli per ottimizzare e riprogettare gli interruttori toehold con prestazioni scarse per scopi diversi, come riportato nel secondo documento.
Il primo modello, basato su una rete neurale convoluzionale (CNN) e perceptron multistrato (MLP), tratta le sequenze toehold come immagini 1D o linee di basi nucleotidiche e identifica schemi di basi e potenziali interazioni tra quelle basi per prevedere buone e cattivi appigli. Il team ha utilizzato questo modello per creare un metodo di ottimizzazione chiamato STORM (Sequence-based Toehold Optimization and Redesign Model), che consente la riprogettazione completa di una sequenza di toehold da zero. Questo strumento “tabula rasa” è ottimale per generare nuovi interruttori di blocco per eseguire una funzione specifica come parte di un circuito genetico sintetico, consentendo la creazione di strumenti biologici complessi.
“La parte davvero interessante di STORM e del modello sottostante è che dopo averlo seminato con i dati di input del primo articolo, siamo stati in grado di mettere a punto il modello con solo 168 campioni e utilizzare il modello migliorato per ottimizzare gli interruttori di appoggio. Ciò mette in discussione l’ipotesi prevalente secondo cui è necessario generare enormi set di dati ogni volta che si desidera applicare un algoritmo di apprendimento automatico a un nuovo problema e suggerisce che l’apprendimento profondo è potenzialmente più applicabile per i biologi sintetici di quanto pensassimo “, ha detto il co-first autrice Jackie Valeri, una studentessa laureata al MIT e al Wyss Institute.
Il secondo modello si basa sull’elaborazione del linguaggio naturale (PNL) e tratta ogni sequenza di appoggio dei piedi come una “frase” composta da schemi di “parole”, imparando infine come certe parole vengono messe insieme per formare una frase coerente. “Mi piace pensare a ogni interruttore sull’appiglio come a una poesia haiku: come un haiku, è un arrangiamento molto specifico di frasi all’interno della sua lingua madre – in questo caso, RNA. Stiamo essenzialmente addestrando questo modello per imparare a scrivere un buon haiku fornendogli molti e molti esempi “, ha detto il co-primo autore Pradeep Ramesh, Ph.D., Visiting Postdoctoral Fellow presso il Wyss Institute e Machine Learning Scientist presso Sherlock Biosciences.
Ramesh ei suoi coautori hanno integrato questo modello basato sulla PNL con il modello basato sulla CNN per creare NuSpeak (Nucleic Acid Speech), un approccio di ottimizzazione che ha permesso loro di ridisegnare gli ultimi 9 nucleotidi di un determinato interruttore di presa mantenendo i restanti 21 nucleotidi intatto. Questa tecnica consente la creazione di appigli progettati per rilevare la presenza di sequenze di RNA patogeni specifici e potrebbero essere utilizzati per sviluppare nuovi test diagnostici.
Il team ha convalidato sperimentalmente entrambe queste piattaforme ottimizzando gli interruttori di appoggio progettati per rilevare i frammenti dal genoma virale SARS-CoV-2 . NuSpeak ha migliorato le prestazioni dei sensori in media del 160%, mentre STORM ha creato versioni migliori di quattro sensori di RNA virale SARS-CoV-2 “cattivi” le cui prestazioni sono migliorate fino a 28 volte.
“Un vero vantaggio delle piattaforme STORM e NuSpeak è che consentono di progettare e ottimizzare rapidamente componenti di biologia sintetica, come abbiamo dimostrato con lo sviluppo di sensori di appoggio per una diagnostica COVID-19 “, ha detto la co-autrice Katie Collins, una studente universitario del MIT presso il Wyss Institute che ha lavorato con il professore associato del MIT Timothy Lu, MD, Ph.D., un autore corrispondente del secondo articolo.
“Gli approcci basati sui dati abilitati dal machine learning aprono la porta a sinergie davvero preziose tra informatica e biologia sintetica, e stiamo appena iniziando a grattare la superficie”, ha affermato Diogo Camacho, Ph.D., un autore corrispondente del secondo documento che è uno scienziato senior di bioinformatica e co-responsabile della Predictive BioAnalytics Initiative presso il Wyss Institute. “Forse l’aspetto più importante degli strumenti che abbiamo sviluppato in questi articoli è che sono generalizzabili ad altri tipi di sequenze basate su RNA come promotori inducibili e riboswitch presenti in natura, e quindi possono essere applicati a un’ampia gamma di problemi e opportunità in biotecnologia e medicina. “
Altri autori degli articoli includono il membro della Wyss Core Faculty e il professore di genetica alla HMS George Church, Ph.D .; e gli studenti laureati Wyss e MIT Miguel Alcantar e Bianca Lepe.
“L’intelligenza artificiale è un’onda che sta appena iniziando a influenzare la scienza e l’industria e ha un potenziale incredibile per aiutare a risolvere problemi intrattabili. Le scoperte descritte in questi studi dimostrano il potere di fondere il calcolo con la biologia sintetica al banco per sviluppare nuove e più potenti tecnologie bioispirate, oltre a portare a nuove intuizioni sui meccanismi fondamentali del controllo biologico “, ha affermato Don Ingber, MD, Ph. D., Direttore fondatore del Wyss Institute. Ingber è anche il Judah Folkman Professor of Vascular Biology presso la Harvard Medical School e il Vascular Biology Program presso il Boston Children’s Hospital, nonché Professore di Bioingegneria presso la John A. Paulson School of Engineering and Applied Sciences di Harvard.
Riferimenti:
7 ottobre 2020, Nature Communications .